Обои оптом со склада, «РЕГИОНПРОЕКТ» надёжный поставщик обоев
Надежный партнер
магазинов обоев
Продаем обои оптом и помогаем
партнерам вести бизнес эффективно
Меры поддержки
клиентов
Умные системы для обойного бизнеса : работа с персоналом, IT — решения, маркетинг, управление ассортиментом
Наша продукция
КОЛЛЕКЦИЯ: Градиент
КОЛЛЕКЦИЯ: Уран
КОЛЛЕКЦИЯ: Шарлотта
КОЛЛЕКЦИЯ: Голден
Весь каталог
Можно просто работать …
А можно работать с максимальной эффективностью, сокращать издержки и растить прибыль.
Для этого нужна система.
У нас 11 магазинов обоев и проверенная умная система управления этими магазинами.
Получить консультацию
Что входит в систему
Школа
профессиональных
продавцов
Видео
Школа профессиональных продавцов OBOI.RU — это специальный проект по подготовке профессиональных продавцов-консультантов для обойных магазинов.
Если ваши продавцы:
- теряют клиентов и продажи
- работают «по наитию», без знаний техник продаж и правил общения с покупателями
- просто просиживают смену и не заинтересованы в результатах работы
то Школа — та самая таблетка, которая поможет увеличить эффективность работы продавцов и выведет продажи на новый уровень.
Качественная работа с продавцами актуальна как для действующих магазинов с полным штатом, так и для новых торговых точек. Продавец — это главная точка контакта вашего бизнеса с покупателем, поэтому их надо:
- правильно искать
- правильно нанимать
- правильно обучать
- правильно проверять знания и умения
Стоит ли инвестировать в найм и обучение продавцов? Какие гарантии, что это окупится? Чем поможет «Школа продавцов»? Посмотрите короткое видео, в котором директор OBOI.RU отвечает на эти и другие вопросы.
Оставьте заявку на бесплатную консультацию по ценам и условиям для наших партнеров
IT — платформа СУП
специализированная умная программа контроля бизнес-показателей для принятия эффективных решений по развитию бизнеса
Она систематизирует данные из вашей учетной системы (1С, Мой склад и т. д.), структурирует и представляет наглядно.
д.), структурирует и представляет наглядно.
Все отчеты и показатели вы видите в онлайн-режиме, а не только когда закрыли отчет по месяцу. Поэтому результаты всех решений видно сразу и их можно оперативно поменять или, например, усилить.
2.1
Блок управления
ассортиментом
Какие задачи поможет решить:
- Определить какие коллекции и артикулы работают максимально эффективно, а какие — наоборот
- Решить какие коллекции заводить в ассортимент, а какие вероятнее всего «зависнут»
- Понять, нужно ли делать распродажи, скидки и акции
Делает полный анализ ассортимента и продаж по артикулу или коллекции. Наглядная система дашбордов с показателями глубины ассортимента, оборачиваемости и степени “здоровья” магазина.
Блок работы с ассортиментом помогает принимать решения на основе данных, а не субъективного, пусть и экспертного, мнения.
2.2
Интерфейс продавца
Какие задачи поможет решить:
- Вся информация, нужная для работы продавцам, в одном месте и в близком доступе
- Снижается многократно количество вопросов руководителю
- Растет лояльность продавцов к работодателю, потому что система мотивации представлена прозрачно и понятно
Удобный интерфейс с доступом к графику работы, расчету ЗП, обучающему разделу. Снимает множество вопросов продавцов, увеличивает лояльность к работодателю за счет прозрачности работы и экономит время руководителя.
2.3
Блок управления маркетингом
Главные показатели работы маркетинга на одном экране.
Какие задачи поможет решить:
- Контролировать эффективность маркетинговых активностей
- Планировать и контролировать бюджет на рекламу и маркетинг
- Отслеживать индекс лояльности, посещаемость магазинов, конверсии и т.
 д.
д.
Система будет работать корректно при полной интеграции с вашей учетной программой. Вы сами решаете какие данные будете передавать на платформу.
Боссборд
Какие задачи поможет решить:
- Контролировать основные бизнес-показатели можно онлайн, с любого устройства
- Быстро увидеть, отклонения от нормы и при необходимости принять рабочее решение
- Быстрый доступ к основной информации: графики, планы, адреса и телефоны и т.д.
Комплексная информация для руководителя с основными коммерческими показателями работы. Все бизнес-показатели онлайн на экране любого устройства.
Зачем вам умная система управления
1
Объективные решения на основе данных
2
Сокращение времени на операционку
3
Оперативный контроль в онлайн-режиме, решения можно принять быстро
4
Рост бизнес — KPI
Как умные системы помогают вести бизнес
кейсы
до 3%
Сократили возвраты по магазинам
на 150%
увеличили количество повторных покупок
92%
NPS (индекс лояльности потребителей) по нашей сети
о компании
Надежное партнерство — важнее всего!
Слово генерального директора
Наша миссия — помочь клиентам развивать бизнес.
РЕГИОНПРОЕКТ — не просто компания, которая отгружает обои со склада. Это в первую очередь помощник, знающий как решить задачи и боли клиентов.
Мы работаем на бизнес наших партнеров, ведь без их роста мы сами застынем на одном месте. А для этого нужно быть надежным поставщиком. К этому мы стремимся и такими являемся.
Компания РЕГИОНПРОЕКТ на рынке с 2006 года и за это время мы постоянно делаем больше, чем от нас ожидают. Мы хотим, чтобы наши клиенты развивались максимально быстро и эффективно.
Генеральный директор,
Юрий Владимирович Киселев
Наш youtube — канал “Настенный бизнес”
Отправить заявку
Разновидности стыковок при поклейке обоев: прямая, смещенная и свободная
Поклейка обоев для многих оптимальный вариант для ремонта комнаты. Для получения достойного результата, необходимо проводить процесс правильно. Внешний вид материала будет смотреться привлекательнее, если не будет шовных зон и других изъянов. Поэтому часто выбирается прямая стыковка обоев, которая требует от мастера внимательности и умения. В статье будет подробно описана прямая стыковка обоев, что это значит, как производиться.
Что это такое
Обойные полотна существуют разные, по данной причине материал остается востребованным длительный период. Клеить обои можно своими руками, что существенно экономит семейный бюджет, но для получения качественного результата нужно знать, как правильно стыковать обои.
Обычно оптимальным вариантом является соединение полотен встык, стыковка рисунка 64, что это значит, спрашивают многие новички. Речь идет о работе с популярной шириной обоев в 64 сантиметра, или шагом узора через данное расстояние. Это стандартный размер и чаще всего работать приходится именно с данным типом полотен.
От выбранного метода, как стыковать обои зависит сложность всего процесса. Если собираются клеить обои стык в стык, то это означает приклеивание их вплотную друг к другу, не оставляя щелей. Вариант нелегкий, дающий оптимальный результат по эстетической красоте отделки.
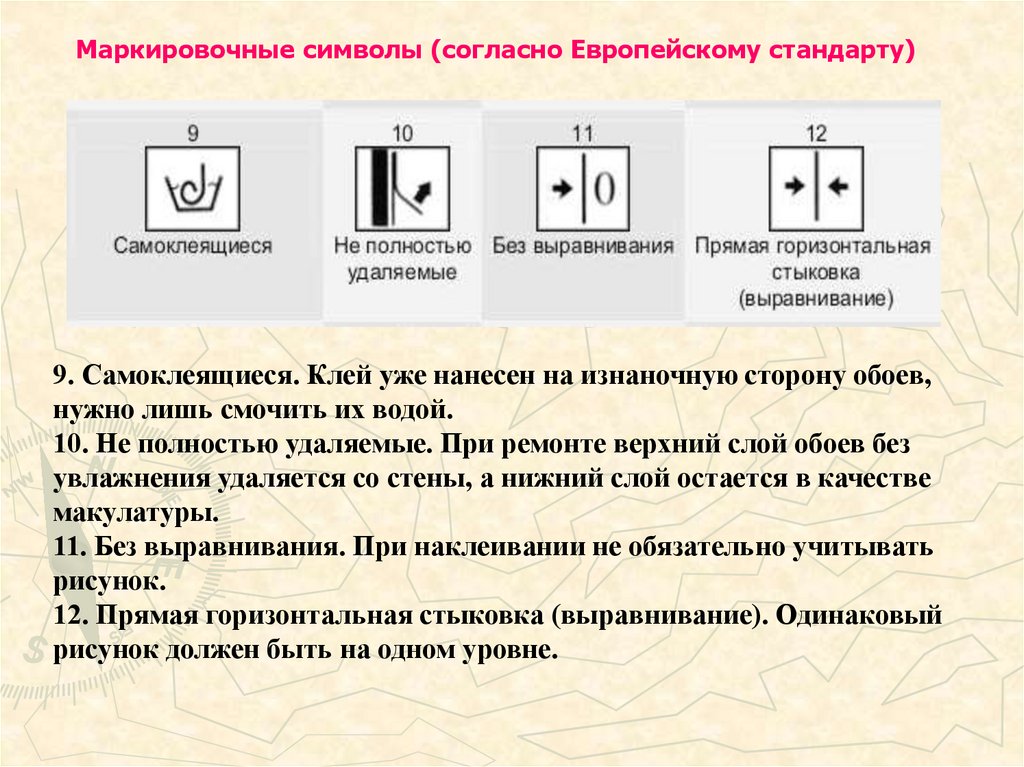
Расшифровка маркировки на обоях
На обойных полотнах есть особые обозначения в виде букв, символов, которые помогают покупателю понять, какой тип полотен перед ним. Знание их значений дает понимание, для каких помещений они подходят, как за ними нужно будет ухаживать, какие свойства характерны. Так существуют следующие маркировочные разделения обоев:
- По составным элементам;
- По специфике поклейки;
- По светоустойчивости;
- По водостойкости;
- По специфике снятия.

Нижеприведенная буквенная маркировка говорит о материалах, из которых производились полотна:
- А- акриловый;
- Б – бумажный;
- ВВ – вспененный винил;
- ПВ – плоский винил;
- РВ – рельефный тип винила;
- ТКС – текстиль;
- СТР – структурные, которые следует после красить;
- СТЛ – стеклообойный тип.
На обойных полотнах есть особые обозначения в виде букв, символов, которые помогают покупателю понять, какой тип полотен перед ним.
Обозначения на обоях — стыковка рисунка
Существует отдельный вариант символа, показывающий, как требуется совместить обои на стене при поклейке. Символами выбран узор стрелками, в зависимости от их направления определяется метод приклеивания рисунка. Бывают нижеописанные варианты:
- Без стыковки, материал может быть поклеен свободно, нет необходимости следить за совмещением узоров;
- Соединение на 1-м уровне. Требуется приклеивать полотна друг к другу на 1-м уровне, то есть когда рисунок соединяется через определённый отрезок.
 Именно это ответ на вопрос: стыковка рисунка на обоях 64 что значит. 64 это через, сколько сантиметров рисунок повторяется;
Именно это ответ на вопрос: стыковка рисунка на обоях 64 что значит. 64 это через, сколько сантиметров рисунок повторяется; - Ступенчатое приклеивание, к приклеенному полотну новое крепиться наполовину выше;
- Встречная поклейка обозначается стрелками, направленными в разные стороны, тогда каждая полоса приклеивается с разворотом на 180 градусов;
- Прямая наклейка, стрелка смотрит прямо. Тогда клеить нужно строго в 1-м направление;
- Может использоваться вариант, когда следует точно сместить полотна при наклеивании. Тогда числителем берется высота узора, заменителем значение смещения обоев.
Существует отдельный вариант символа, показывающий, как требуется совместить обои на стене при поклейке.
Разновидности стыковок
Обычно обои делаются не только разными по цвету, но и по узору. Рисунки делаются с повтором через конкретное расстояние, это значение называют раппортом. И есть различные виды наклеивания полотен, исходя из этого параметра.
Рисунки делаются с повтором через конкретное расстояние, это значение называют раппортом.
Прямая стыковка
Данная технология выбирается, когда узоры в каждом рулоне в начале полосы имеют одинаковый вид, и повторение идет равномерно по всей длине. Этот тип обозначается 2-мя стрелами, которые направляются друг к другу. Рядом прописывается цифра, значащая через, сколько предполагается повторение узора. По этой цифре вычисляют, какой длины полосы нужно будет резать, чтобы получилось состыковать узоры идеально.
Данная технология выбирается, когда узоры в каждом рулоне в начале полосы имеют одинаковый вид, и повторение идет равномерно по всей длине.
Смещенная
Данный вид прописывается, когда на полотнах изображены геометрические элементы. Используются символы в виде двух стрелок, смотрящих друг на друга, но одна стоит чуть ниже к другой. Тогда в разных рулонах узор начинается с различных фрагментов. При поклейке обои смещается на пол раппорта, указанного на упаковке.
Для уменьшения выброса, специалисты рекомендуют нарезать полосы из двух разных рулонов.
Данный вид прописывается, когда на полотнах изображены геометрические элементы.
Свободная
В данном случае рисунки не имеют определённого порядка, являются хаотичным изображением. Тогда не требуется при нарезке полос следить за раппортом, его не прописывают. Можно будет клеить полотна просто друг за другом без состыковки элементов.
Можно клеить полотна просто друг за другом без состыковки элементов.
Встречная
Подходящий вариант для разнообразных узоров. Рисуется на упаковке знак из стрелок, которые направлены каждая в свою сторону, противоположную от другой. Каждая новая полоса клеиться противоположно предыдущей.
Подходящий вариант для разнообразных узоров.
Смешанная
В данном случае можно будет использовать при ремонтных работах разные варианты приклеивания. Основная площадь приклеивается одним из выбранных типов поклейки, а труднодоступные зоны, такие как углы, можно будет клеить более легким вариантом, чтобы ускорить и упростить процесс.
В данном случае можно будет использовать при ремонтных работах разные варианты приклеивания.
Плюсы и минусы разных видов стыковки
Если сравнивать варианты поклейки встык, то они выбираются по типу изображения, и тогда нужно учитывать их особенности уже при выборе обоев. Самый простой в выполнении метод это свободная поклейка, с ней без труда справится и новичок.
Прямой и смещенный вариант предполагает выброс части обоев, что может повысить их расход. Но часто сами узоры отличаются большей привлекательностью.
От длины раппорта зависит размер выброса, чем он меньше, тем больше полотен придется выбросить.
Если же выбирать между работой встык или внахлест, то второй способ больше бросается в глаза, и обычно используется лишь для работы на проблемных углах. Если выбран толстый тип обоев, то результат может стать слишком заметным и неопрятным.
Если сравнивать варианты поклейки встык, то они выбираются по типу изображения.
Полезные советы
Многие предпочитают клеить обои самостоятельно. Полезно, знать хитрости профессионалов, помогающие понять, как клеить обои встык без щелей. Выделяются нижеперечисленные принципы работы:
Полезно, знать хитрости профессионалов, помогающие понять, как клеить обои встык без щелей. Выделяются нижеперечисленные принципы работы:
- Для того чтобы края не сгибались и не загибалась можно применить специальную ленту для поклейки обоев на стыках;
- Краевую область полос необходимо хорошо промазать клеевым раствором;
- После соединения полотна к стене сверху прокатывается валик из резины, для упрочнения сцепки;
- При приклеивании не стоит растягивать полотна, момент приводит к получению по окончанию просушки отклеивающихся участков.
После соединения полотна к стене сверху прокатывается валик из резины, для упрочнения сцепки.
Приклеивание обойных полотен будет проходить проще, если заранее при выборе материала будет изучена его маркировка, тогда мастер заранее будет знать, какой метод работы нужно будет выполнить. При соблюдении раппорта без труда получают единый рисунок на стенах, радующий владельцев комнаты.
Видео: Как клеить стыки обоев
Стыковка фигур на рабочем столе :: Свойства фигуры (графика)
Стыковка фигур на рабочем столе :: Свойства фигуры (графика)| Графика |
Закрепление фигур на рабочем столе
Вы можете закрепить фигуры на рабочем столе MATLAB, щелкнув кнопку стыковки, которая появляется в правом конце строки меню. После пристыковки фигурки помещаются в контейнер группы фигурок, который вы также можете пристыковать и отстыковать.
Вы можете выбрать любое расположение фигурок в контейнере. На следующем рисунке показано, как выбрать различные варианты расположения фигур. После закрепления контейнер фигуры отображает панель инструментов и строку меню фигуры с фокусом.
Свойства фигуры, влияющие на стыковку
Есть два свойства фигуры, которые связаны со стыковкой фигуры – DockControls и WindowStyle .
DockControls
Свойство DockControls управляет отображением элементов управления, используемых для закрепления фигур. Установка для DockControls значения off удаляет кнопку стыковки из строки меню и отключает стыковку из меню рисунка Desktop .
WindowStyle
Когда вы устанавливаете свойство WindowStyle на пристыковано , MATLAB закрепляет фигуру на рабочем столе в контейнере группы фигур.
Если для WindowStyle установлено значение , закрепленное ,
- MATLAB автоматически устанавливает
DockControlsнана. - Нельзя установить для свойства
DockControlsзначениеот. - Невозможно установить свойство фигуры
Позиция.
Стыковка фигур автоматически
Если вы хотите, чтобы MATLAB всегда стыковал фигуры, установите значение по умолчанию Свойство WindowStyle до пристыковано к . Следующее утверждение,
Следующее утверждение,
создает значение по умолчанию для свойства WindowStyle на корневом уровне. Выполнение этого оператора в командной строке устанавливает WindowStyle всех фигур на время вашего сеанса MATLAB (если вы не измените значение).
Поместите этот оператор в файл startup.m , чтобы MATLAB всегда закреплял фигуры. См. запуск для получения дополнительной информации о запуск.м .
Создание нестыкуемой фигуры
В тех случаях, когда вы не хотите, чтобы пользователи могли закреплять фигуры (например, фигуры, используемые для графических интерфейсов), вы должны установить следующие свойства фигуры:
-
DockControls отдовыкл. -
WindowStyle отдообычныйилимодальный -
HandleVisibility отдовыключеноилиобратный вызов
| Фигура Объекты | Позиционирование Фигур |
Молекулярная стыковка: мощный подход к открытию лекарств на основе структуры
[1] Jorgensen WL.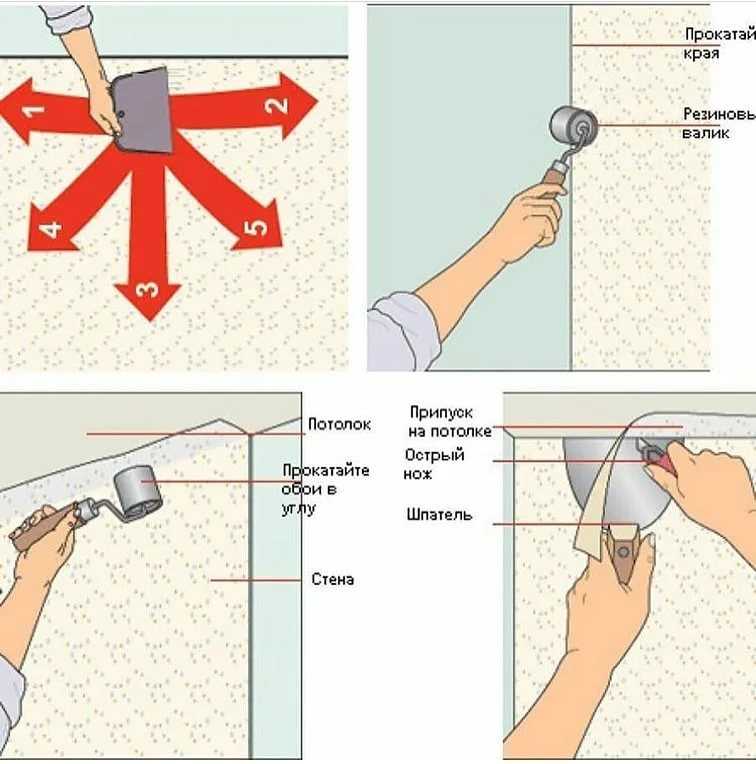 Многочисленные роли вычислений в открытии лекарств. Наука. 2004;303(5665):1813–1818. [PubMed] [Google Scholar]
Многочисленные роли вычислений в открытии лекарств. Наука. 2004;303(5665):1813–1818. [PubMed] [Google Scholar]
[2] Баджорат Дж. Интеграция виртуального и высокопроизводительного скрининга. Nat Rev Drug Discov. 2002; 1(11):882–89.4. [PubMed] [Google Scholar]
[3] Walters WP, Stahl MT, Murcko MA. Виртуальный скрининг - обзор. Препарат Дисков. Сегодня. 1998; 3: 160–178. [Google Scholar]
[4] Лангер Т., Хоффманн Р.Д. Виртуальный скрининг: эффективный инструмент для выявления структуры потенциальных клиентов? Курр Фарм Дез. 2001;7(7):509–527. [PubMed] [Google Scholar]
[5] Kitchen DB, Decornez H, Furr JR, Bajorath J. Стыковка и оценка в виртуальном скрининге для обнаружения наркотиков: методы и приложения. Nat Rev Drug Discov. 2004;3(11):935–949. [PubMed] [Google Scholar]
[6] Gohlke H, Klebe G. Подходы к описанию и прогнозированию аффинности связывания низкомолекулярных лигандов с макромолекулярными рецепторами. Angew Chem Int Ed Engl. 2002;41(15):2644–2676.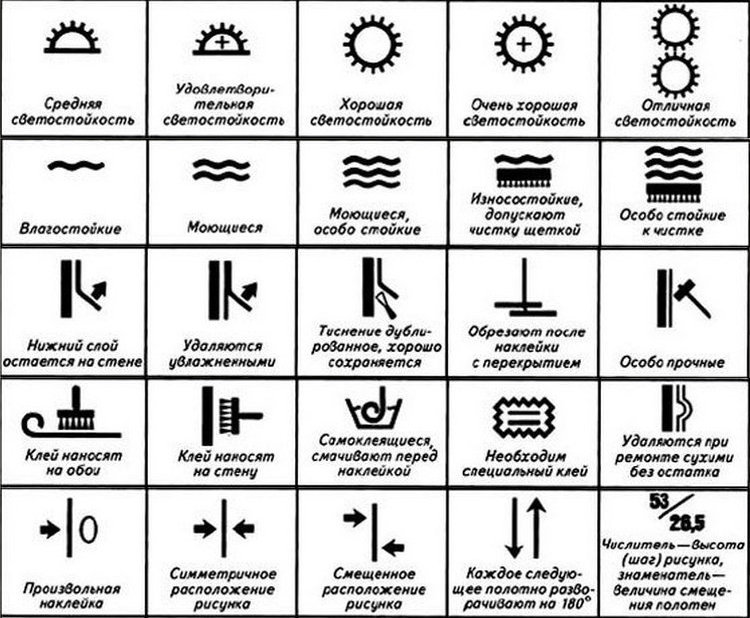 [PubMed] [Google Scholar]
[PubMed] [Google Scholar]
[7] Moitessier N, Englebienne P, Lee D, Lawandi J, Corbeil CR. На пути к разработке универсальных, быстрых и высокоточных методов стыковки/скоринга: долгий путь. Бр Дж. Фармакол. 2008; 153 (Приложение 1): S7–26. [Бесплатная статья PMC] [PubMed] [Google Scholar]
[8] Шойхет Б.К., Макговерн С.Л., Вэй Б., Ирвин Дж.Дж. Хиты, лиды и артефакты виртуального и высокопроизводительного скрининга. 2002. Молекулярная информатика: противостояние сложности. [Google Scholar]
[9] Бэйли Д., Браун Д. Высокопроизводительная химия и структурный дизайн: выживает самый умный. Наркотиков Дисков сегодня. 2001;6(2):57–59. [PubMed] [Google Scholar]
[10] Kuntz ID, Blaney JM, Oatley SJ, Langridge R, Ferrin TE. Геометрический подход к взаимодействиям макромолекула-лиганд. Дж Мол Биол. 1982;161(2):269–288. [PubMed] [Google Scholar]
[11] Гальперин И., Ма Б., Вольфсон Х., Нусинов Р. Принципы стыковки: обзор алгоритмов поиска и руководство по скоринговым функциям. Белки. 2002;47(4):409–443. [PubMed] [Google Scholar]
Белки. 2002;47(4):409–443. [PubMed] [Google Scholar]
[12] Coupez B, Lewis RA. Стыковка и подсчет очков — теоретически легко, практически невозможно? Курр Мед Хим. 2006;13(25):2995–3003. [PubMed] [Google Scholar]
[13] Контойанни М., Мадхав П., Суханек Э., Сейбел В. Теоретические и практические аспекты виртуального скрининга: проторенное поле? Курр Мед Хим. 2008;15(2):107–116. [PubMed] [Академия Google]
[14] Brooijmans N, Kuntz ID. Алгоритмы молекулярного распознавания и докинга. Annu Rev Biophys Biomol Struct. 2003; 32: 335–373. [PubMed] [Google Scholar]
[15] тен Бринк Т., Экснер Т.Э. Влияние протонирования, таутомерных и стереоизомерных состояний на результаты докинга белок-лиганд. Модель J Chem Inf. 2009;49(6):1535–1546. [PubMed] [Google Scholar]
[16] Cross JB, Thompson DC, Rai BK, Baber JC, Fan KY, Hu Y, Humblet C. Сравнение нескольких программ молекулярной стыковки: предсказание позы и точность виртуального скрининга. Модель J Chem Inf. 2009 г. ;49(6):1455–1474. [PubMed] [Google Scholar]
;49(6):1455–1474. [PubMed] [Google Scholar]
[17] Li X, Li Y, Cheng T, Liu Z, Wang R. Оценка эффективности четырех программ молекулярной стыковки на разнообразном наборе белково-лигандных комплексов. J Comput Chem. 2010;31(11):2109–2125. [PubMed] [Google Scholar]
[18] Plewczynski D, Lazniewski M, Augustyniak R, Ginalski K. Можно ли доверять результатам стыковки? Оценка семи часто используемых программ в базе данных PDBbind. J Comput Chem. Doi 2010: 10.1002/jcc.21643 [PubMed] [Google Scholar]
[19] Макконки Б.Дж., Соболев В., Эдельман М. Эффективность современных методов стыковки лиганд-белок. Современная наука. 2002; 83: 845–855. [Google Scholar]
[20] Гудфорд П.Дж. Вычислительная процедура для определения энергетически выгодных мест связывания на биологически важных макромолекулах. J Med Chem. 1985; 28(7):849–857. [PubMed] [Google Scholar]
[21] Kastenholz MA, Pastor M, Cruciani G, Haaksma EE, Fox T. GRID/CPCA: новый вычислительный инструмент для разработки селективных лигандов. J Med Chem. 2000;43(16):3033–3044. [PubMed] [Академия Google]
J Med Chem. 2000;43(16):3033–3044. [PubMed] [Академия Google]
[22] Левитт Д.Г., Банашак Л.Дж. POCKET: метод компьютерной графики для идентификации и отображения белковых полостей и окружающих их аминокислот. Дж Мол График. 1992;10(4):229–234. [PubMed] [Google Scholar]
[23] Ласковски Р.А. SURFNET: программа для визуализации молекулярных поверхностей, полостей и межмолекулярных взаимодействий. Дж Мол График. 1995;13(5):323–330. 307–328. [PubMed] [Google Scholar]
[24] Glaser F, Morris RJ, Najmanovich RJ, Laskowski RA, Thornton JM. Метод локализации карманов связывания лигандов в белковых структурах. Белки. 2006;62(2):479–488. [PubMed] [Google Scholar]
[25] Brady GP, Jr., Stouten PF. Быстрое прогнозирование и визуализация карманов связывания белка с помощью PASS. J Comput Aided Mol Des. 2000;14(4):383–401. [PubMed] [Google Scholar]
[26] Мезей М. Новый метод картирования макромолекулярной топографии. Графовая модель J-Mol. 2003;21(5):463–472. [PubMed] [Google Scholar]
[27] Fischer E. Einfluss der configuration auf die wirkung derenzyme. Бер. Дт. хим. Гэс. 1894; 27: 2985–2993. [Академия Google]
Einfluss der configuration auf die wirkung derenzyme. Бер. Дт. хим. Гэс. 1894; 27: 2985–2993. [Академия Google]
[28] Кошланд Д.Е., младший. Корреляция структуры и функции в ферментном действии. Наука. 1963; 142: 1533–1541. [PubMed] [Google Scholar]
[29] Hammes GG. Множественные конформационные изменения при ферментативном катализе. Биохимия. 2002;41(26):8221–8228. [PubMed] [Google Scholar]
[30] Рари М., Крамер Б., Ленгауэр Т., Клебе Г. Быстрый гибкий метод стыковки с использованием алгоритма пошагового построения. Дж Мол Биол. 1996;261(3):470–489. [PubMed] [Google Scholar]
[31] Morris GM, Goodsell DS, Halliday RS, Huey R, Hart WE, Belew RK, Olson AJ. Автоматическая стыковка с использованием генетического алгоритма Ламарка и эмпирической функции свободной энергии связывания. Журнал вычислительной химии. 1998;19(14):1639–1662. [Google Scholar]
[32] Джонс Дж., Уиллетт П., Глен Р.С., Лич А.Р., Тейлор Р. Разработка и проверка генетического алгоритма гибкой стыковки.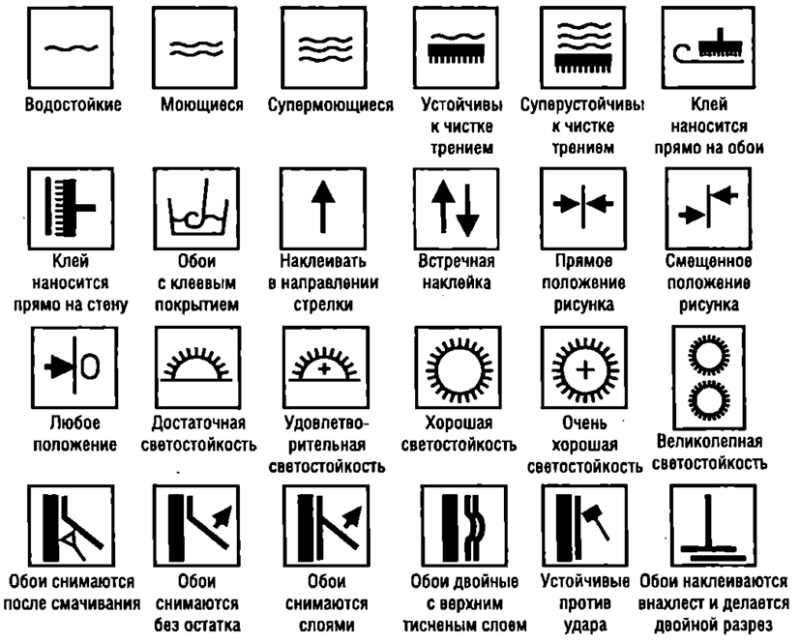 Дж Мол Биол. 1997;267(3):727–748. [PubMed] [Google Scholar]
Дж Мол Биол. 1997;267(3):727–748. [PubMed] [Google Scholar]
[33] Friesner RA, Banks JL, Murphy RB, Halgren TA, Klicic JJ, Mainz DT, Repasky MP, Knoll EH, Shelley M, Perry JK, Shaw DE, Francis P, Shenkin PS . Glide: новый подход к быстрой и точной стыковке и подсчету очков. 1 Способ и оценка точности стыковки. J Med Chem. 2004;47(7):1739–1749. [PubMed] [Google Scholar]
[34] McGann MR, Almond HR, Nicholls A, Grant JA, Brown FK. Гауссовы функции стыковки. Биополимеры. 2003;68(1):76–90. [PubMed] [Google Scholar]
[35] Perola E, Walters WP, Charifson PS. Подробное сравнение текущих методов стыковки и оценки систем, имеющих значение для фармацевтики. Белки. 2004;56(2):235–249. [PubMed] [Google Scholar]
[36] Шерман В., Дэй Т., Джейкобсон М.П., Фризнер Р.А., Фарид Р. Новая процедура моделирования эффектов соответствия, индуцированных лигандом/рецептором. J Med Chem. 2006;49(2): 534–553. [PubMed] [Google Scholar]
[37] Jiang F, Kim SH. «Мягкий докинг»: сопоставление кубов молекулярных поверхностей. Дж Мол Биол. 1991;219(1):79–102. [PubMed] [Google Scholar]
Дж Мол Биол. 1991;219(1):79–102. [PubMed] [Google Scholar]
[38] Claussen H, Buning C, Rarey M, Lengauer T. FlexE: эффективный молекулярный докинг с учетом вариаций структуры белка. Дж Мол Биол. 2001;308(2):377–395. [PubMed] [Google Scholar]
[39] Alonso H, Bliznyuk AA, Gready JE. Сочетание стыковки и молекулярно-динамического моделирования при разработке лекарств. Med Res Rev. 2006;26(5):531–568. [PubMed] [Академия Google]
[40] Sander T, Liljefors T, Balle T. Прогнозирование конформации рецептора для связывания агониста iGluR2: стыковка QM/MM с обширным конформационным ансамблем, созданным с использованием анализа в нормальном режиме. Графовая модель J-Mol. 2008;26(8):1259–1268. [PubMed] [Google Scholar]
[41] Subramanian J, Sharma S, C BR. Новый компьютерный анализ индуцированных лигандом конформационных изменений в сайтах связывания АТФ циклинзависимых киназ. J Med Chem. 2006;49(18):5434–5441. [PubMed] [Академия Google]
[42] Субраманиан Дж., Шарма С. , C BR. Моделирование и выбор гибких белков для разработки лекарств на основе структуры: движение основной и боковой цепи в p38 MAPK. ХимМедХим. 2008;3(2):336–344. [PubMed] [Google Scholar]
, C BR. Моделирование и выбор гибких белков для разработки лекарств на основе структуры: движение основной и боковой цепи в p38 MAPK. ХимМедХим. 2008;3(2):336–344. [PubMed] [Google Scholar]
[43] Бринт А.Т., Уиллетт П. Алгоритмы идентификации трехмерных максимальных общих подструктур. Дж. Хим. Инф. вычисл. науч. 1987; 27: 152–158. [Google Scholar]
[44] Фишер Д., Норел Р., Вольфсон Х., Нуссинов Р. Поверхностные мотивы с помощью метода компьютерного зрения: поиск, обнаружение и последствия для распознавания белок-лиганд. Белки. 1993;16(3):278–292. [PubMed] [Google Scholar]
[45] Норел Р., Фишер Д., Вольфсон Х.Дж., Нуссинов Р. Распознавание молекулярной поверхности с помощью метода компьютерного зрения. Белок англ. 1994;7(1):39–46. [PubMed] [Google Scholar]
[46] Miller MD, Kearsley SK, Underwood DJ, Sheridan RP. FLOG: система для выбора «квазигибких» лигандов, комплементарных рецептору известной трехмерной структуры. J Comput Aided Mol Des. 1994;8(2):153–174. [PubMed] [Google Scholar]
[PubMed] [Google Scholar]
[47] Diller DJ, Merz KM, Jr. Высокопроизводительная стыковка для проектирования библиотек и определения приоритетов библиотек. Белки. 2001;43(2):113–124. [PubMed] [Академия Google]
[48] Буркхард П., Тейлор П., Уолкиншоу, доктор медицины. Пример белкового лиганда, найденного в результате анализа базы данных: описание метода докинга и его проверка с помощью рентгенограммы 2,3 А структуры комплекса тромбин-лиганд. Дж Мол Биол. 1998;277(2):449–466. [PubMed] [Google Scholar]
[49] DesJarlais RL, Sheridan RP, Dixon JS, Kuntz ID, Venkataraghavan R. Стыковка гибких лигандов с макромолекулярными рецепторами по молекулярной форме. J Med Chem. 1986;29(11):2149–2153. [PubMed] [Академия Google]
[50] Кунц И.Д., Лич А.Р. Конформационный анализ гибких лигандов в сайтах макромолекулярных рецепторов. Дж. Вычисл. хим. 1992; 13: 730–748. [Google Scholar]
[51] Ewing TJ, Makino S, Skillman AG, Kuntz ID. DOCK 4.0: стратегии поиска для автоматизированной молекулярной стыковки гибких баз данных молекул. J Comput Aided Mol Des. 2001;15(5):411–428. [PubMed] [Google Scholar]
J Comput Aided Mol Des. 2001;15(5):411–428. [PubMed] [Google Scholar]
[52] Welch W, Ruppert J, Jain AN. Hammerhead: быстрая, полностью автоматизированная стыковка гибких лигандов с сайтами связывания белков. хим. биол. 1996;3(6):449–462. [PubMed] [Google Scholar]
[53] Schnecke V, Kuhn LA. Виртуальный скрининг с сольватацией и лиганд-индуцированной комплементарностью. Перспективы открытия и разработки лекарств. 2000;20:171–190. [Google Scholar]
[54] Жолдос З., Рейд Д., Саймон А., Садджад Б.С., Джонсон А.П. eHiTS: инновационный подход к проблемам функции стыковки и подсчета очков. Curr Protein Pept Sci. 2006;7(5):421–435. [PubMed] [Google Scholar]
[55] Миранкер А., Карплюс М. Функциональные карты сайтов связывания: метод одновременного поиска нескольких копий. Белки. 1991;11(1):29–34. [PubMed] [Google Scholar]
[56] Eisen MB, Wiley DC, Karplus M, Hubbard RE. HOOK: программа для поиска новой молекулярной архитектуры, которая удовлетворяет химическим и стерическим требованиям сайта связывания макромолекулы. Белки. 1994;19(3):199–221. [PubMed] [Google Scholar]
Белки. 1994;19(3):199–221. [PubMed] [Google Scholar]
[57] Bohm HJ. LUDI: основанный на правилах автоматический дизайн новых заместителей для ингибиторов ферментов. J Comput Aided Mol Des. 1992;6(6):593–606. [PubMed] [Google Scholar]
[58] Goodsell DS, Lauble H, Stout CD, Olson AJ. Автоматизированный докинг в кристаллографии: анализ субстратов аконитазы. Белки. 1993;17(1):1–10. [PubMed] [Google Scholar]
[59] Hart TN, Read RJ. Метод многозаходной стыковки Монте-Карло. Белки. 1992;13(3):206–222. [PubMed] [Google Scholar]
[60] Goodsell DS, Olson AJ. Автоматическая стыковка подложек с белками путем имитации отжига. Белки. 1990;8(3):195–202. [PubMed] [Google Scholar]
[61] Абагян Р., Тотров М., Кузнецов Д. ICM-Новый метод моделирования и дизайна белков: приложения для стыковки и прогнозирования структуры на основе искаженной нативной конформации. Дж. Вычисл. хим. 1994;15:488–506. [Google Scholar]
[62] McMartin C, Bohacek RS. QXP: мощные, быстрые компьютерные алгоритмы для разработки лекарств на основе структуры. J Comput Aided Mol Des. 1997;11(4):333–344. [PubMed] [Google Scholar]
J Comput Aided Mol Des. 1997;11(4):333–344. [PubMed] [Google Scholar]
[63]
Accelrys Inc., Сан-Диего, Калифорния, США.
[64] Оширо К.М., Кунц И.Д., Диксон Дж.С. Гибкая стыковка лигандов с использованием генетического алгоритма. J Comput Aided Mol Des. 1995;9(2):113–130. [PubMed] [Google Scholar]
[65] Verdonk ML, Cole JC, Hartshorn MJ, Murray CW, Taylor RD. Улучшенная стыковка белок-лиганд с использованием GOLD. Белки. 2003;52(4):609–623. [PubMed] [Google Scholar]
[66] Кларк К.П. Ajay, Гибкая стыковка лигандов без регулировки параметров в четырех комплексах лиганд-рецептор. J Comput Chem. 1995; 16:1210–1226. [Google Scholar]
[67] Taylor JS, Burnett RM. DARWIN: программа для стыковки гибких молекул. Белки. 2000;41(2):173–191. [PubMed] [Google Scholar]
[68] Cornell WD, Cieplak P, Bayly CI, Gould IR, Merz KM, Ferguson DM, Spellmeyer DC, Fox T, Caldwell JW, Kollman PA. Силовое поле второго поколения для моделирования белков, нуклеиновых кислот и органических молекул. Варенье. хим. соц. 1995;117:5179–5197. [Google Scholar]
Варенье. хим. соц. 1995;117:5179–5197. [Google Scholar]
[69] Weiner SJ, Kollman PA, Case DA, Singh UC, Ghio C, Alagona G, Profeta S, Jr., Weiner P. Новое силовое поле для молекулярно-механического моделирования нуклеиновых кислот и белков. Варенье. хим. соц. 1984; 106: 765–784. [Google Scholar]
[70] Брукс Б.Р., Брукколери Р.Е., Олафсон Б.Д., Стейтс Д.Дж., Сваминатан С., Карплюс М. CHARMM: программа для расчета макромолекулярной энергии, минимизации и динамики. Дж. Вычисл. хим. 1983; 4: 187–217. [Академия Google]
[71] Коллман П.А. Расчеты свободной энергии: приложения к химическим и биохимическим явлениям. хим. 1993; 93: 2395–2417. [Google Scholar]
[72] Аквист Дж., Лужков В.Б., Брандсдал Б.О. Аффинность связывания лиганда из моделирования МД. Acc Chem Res. 2002;35(6):358–365. [PubMed] [Google Scholar]
[73] Carlson HA, Jorgensen WL. Метод расширенного линейного отклика для определения свободных энергий гидратации. J Phys Chem. 1995;99:10667–10673. [Google Scholar]
[Google Scholar]
[74] Шойхет Б.К., Страуд Р.М., Санти Д.В., Кунц И.Д., Перри К.М. Основанное на структуре открытие ингибиторов тимидилатсинтазы. Наука. 1993;259(5100):1445–1450. [PubMed] [Google Scholar]
[75] Мишель Дж., Вердонк М.Л., Эссекс Дж.В. Прогнозирование сродства связывания белок-лиганд с помощью имплицитного моделирования растворителя: инструмент для оптимизации свинца? J Med Chem. 2006;49(25):7427–7439. [PubMed] [Google Scholar]
[76] Briggs JM, Marrone TJ, McCammon JA. Новые горизонты вычислительной науки и актуальность для фармацевтического дизайна. Тенденции Кардиовасц. Мед. 1996; 6: 198–206. [PubMed] [Google Scholar]
[77] Bohm HJ. Прогнозирование констант связывания белковых лигандов: быстрый метод приоритизации совпадений, полученных в результате проектирования de novo или программ поиска в базе данных 3D. J Comput Aided Mol Des. 1998;12(4):309–323. [PubMed] [Google Scholar]
[78] Гельхаар Д.К., Верховкер Г.М., Рейто П.А., Шерман С. Дж., Фогель Д.Б., Фогель Л.Дж., Фрир С.Т. Молекулярное распознавание ингибитора AG-1343 протеазой ВИЧ-1: конформационно гибкая стыковка путем эволюционного программирования. хим. биол. 1995;2(5):317–324. [PubMed] [Google Scholar]
Дж., Фогель Д.Б., Фогель Л.Дж., Фрир С.Т. Молекулярное распознавание ингибитора AG-1343 протеазой ВИЧ-1: конформационно гибкая стыковка путем эволюционного программирования. хим. биол. 1995;2(5):317–324. [PubMed] [Google Scholar]
[79] Верховкер Г.М., Бузида Д., Гельхаар Д.К., Рейто П.А., Артурс С., Колсон А.Б., Фрир С.Т., Ларсон В., Люти Б.А., Маррон Т., Роуз П.В. Расшифровка распространенных сбоев при молекулярном докинге лиганд-белковых комплексов. J Comput Aided Mol Des. 2000;14(8):731–751. [PubMed] [Академия Google]
[80] Джейн А.Н. Подсчет нековалентных взаимодействий белок-лиганд: непрерывная дифференцируемая функция, настроенная для вычисления аффинности связывания. J Comput Aided Mol Des. 1996;10(5):427–440. [PubMed] [Google Scholar]
[81] Head RD, Smythe ML, Oprea TI, Waller CL, Green SM, Marshall GR. ПРОВЕРКА: новый метод предсказания связывания новых лигандов на основе рецепторов. Варенье. хим. соц. 1996; 118:3959–3969. [Google Scholar]
[82] Gehlhaar DK, Moerder KE, Zichi D, Sherman CJ, Ogden RC, Freer ST. Дизайн ингибиторов ферментов de novo путем генерации лигандов Монте-Карло. J Med Chem. 1995;38(3):466–472. [PubMed] [Google Scholar]
Дизайн ингибиторов ферментов de novo путем генерации лигандов Монте-Карло. J Med Chem. 1995;38(3):466–472. [PubMed] [Google Scholar]
[83] Элдридж, М.Д., Мюррей К.В., Аутон Т.Р., Паолини Г.В., Ми Р.П. Эмпирические оценочные функции: I. Разработка быстрой эмпирической оценочной функции для оценки аффинности связывания лигандов в рецепторных комплексах. J Comput Aided Mol Des. 1997;11(5):425–445. [PubMed] [Google Scholar]
[84] Muegge I, Martin YC. Общая и быстрая функция оценки взаимодействий белок-лиганд: упрощенный потенциальный подход. J Med Chem. 1999;42(5):791–804. [PubMed] [Google Scholar]
[85] Mitchell JBO, Laskowski RA, Alex A, Thornton JM. Пип-потенциал средней силы, описывающий белок-лигандные взаимодействия: I. генерирующий потенциал. Дж. Вычисл. хим. 1999;20(11):1165–1176. [Google Scholar]
[86] Ищенко А.В., Шахнович Е.И. SMall Molecule Growth 2001 (SMoG2001): улучшенная функция оценки взаимодействия белок-лиганд, основанная на знаниях. J Med Chem. 2002;45(13):2770–2780. [PubMed] [Google Scholar]
2002;45(13):2770–2780. [PubMed] [Google Scholar]
[87] Feher M, Deretey E, Roy S. BHB: простая функция оценки, основанная на знаниях, для повышения эффективности проверки базы данных. J Chem Inf Comput Sci. 2003;43(4):1316–1327. [PubMed] [Академия Google]
[88] Верховкер Г., Аппельт К., Фрир С.Т., Виллафранка Ю.Е. Эмпирические расчеты свободной энергии лиганд-белковых кристаллографических комплексов. I. Основанные на знаниях потенциалы взаимодействия лиганд-белок, применяемые для предсказания аффинности связывания протеазы вируса иммунодефицита человека 1. Белок англ. 1995;8(7):677–691. [PubMed] [Google Scholar]
[89] Wallqvist A, Jernigan RL, Covell DG. Параметризация свободной энергии связывания фермента с ингибитором на основе предпочтений. Приложения к дизайну ингибитора протеазы ВИЧ-1. Белковая наука. 1995;4(9):1881–1903. [Бесплатная статья PMC] [PubMed] [Google Scholar]
[90] Gohlke H, Hendlich M, Klebe G. Функция оценки, основанная на знаниях, для прогнозирования взаимодействий белок-лиганд. Дж Мол Биол. 2000;295(2):337–356. [PubMed] [Google Scholar]
Дж Мол Биол. 2000;295(2):337–356. [PubMed] [Google Scholar]
[91] ДеВитте Р.С., Шахнович Э.И. SMoG: метод проектирования de Novo, основанный на простых, быстрых и точных оценках свободной энергии. 1 Методология и подтверждающие данные. Варенье. хим. соц. 1996; 118:11733–11744. [Google Scholar]
[92] Чарифсон П.С., Коркери Дж.Дж., Мурко М.А., Уолтерс В.П. Консенсусная оценка: метод повышения частоты попаданий из баз данных стыковки трехмерных структур с белками. J Med Chem. 1999;42(25):5100–5109. [PubMed] [Google Scholar]
[93] Feher M. Согласованная оценка взаимодействий белок-лиганд. Наркотиков Дисков сегодня. 2006;11(9-10):421–428. [PubMed] [Google Scholar]
[94] Кларк Р.Д., Стрижев А., Леонард Дж.М., Блейк Дж.Ф., Мэтью Дж.Б. Согласованная оценка взаимодействий лиганд/белок. Графовая модель J-Mol. 2002;20(4):281–295. [PubMed] [Google Scholar]
[95] Сринивасан Дж., Читэм Т.Е., Чиплак П., Коллман П.А., Дело Д.А. Исследования стабильности спиралей ДНК, РНК и фосфорамидат-ДНК с помощью растворителя. Журнал Американского химического общества. 1998;120(37):9401–9409. [Google Scholar]
Журнал Американского химического общества. 1998;120(37):9401–9409. [Google Scholar]
[96] Коллман П.А., Массова И., Рейес С., Кун Б., Хуо С., Чонг Л., Ли М., Ли Т., Дуан И., Ван В., Донини О., Чеплак П., Шринивасан Дж., Кейс Д.А. , Cheatham TE., 3rd Расчет структур и свободных энергий сложных молекул: сочетание молекулярной механики и моделей сплошных сред. Acc Chem Res. 2000;33(12):889–897. [PubMed] [Google Scholar]
[97] Still WC, Tempczyk A, Hawley RC, Hendrickson T. Semianalytical Treatment of Solvation for Molecular Mechanics and Dynamics. Варенье. хим. соц. 1990;112(16):6127–6129. [Google Scholar]
[98] Guimaraes CR, Mathiowetz AM. Устранение ограничений с помощью процедуры оценки MM-GB/SA с использованием метода WaterMap и расчетов возмущения свободной энергии. Модель J Chem Inf. 50 (4): 547–559. [PubMed] [Google Scholar]
[99] Сингх Н., Уоршел А. Расчеты абсолютной свободной энергии связывания: точность вычислительной оценки взаимодействий белок-лиганд. Белки. 2010;78(7):1705–1723. [Бесплатная статья PMC] [PubMed] [Google Scholar]
Белки. 2010;78(7):1705–1723. [Бесплатная статья PMC] [PubMed] [Google Scholar]
[100] Гэбб Х.А., Джексон Р.М., Штернберг М.Дж. Моделирование стыковки белков с использованием комплементарности формы, электростатики и биохимической информации. Дж Мол Биол. 1997;272(1):106–120. [PubMed] [Google Scholar]
[101] Брон С., Кербош Дж. Алгоритм 457: поиск всех клик неориентированного графа. Коммуникации АКМ. 1973;16(9):575–576. [Google Scholar]
[102] Meng EC, Shoichet BK, Kuntz ID. Автоматическая стыковка с оценкой энергии на основе сети. Дж. Вычисл. хим. 1992; 13: 505–524. [Академия Google]
[103] Zou XQ, Sun Y, Kuntz ID. Включение сольватации в расчеты свободной энергии связывания лиганда с использованием обобщенной модели Борна. Варенье. хим. соц. 1999; 121:8033–8043. [Google Scholar]
[104] Тротт О., Олсон А.Дж. AutoDock Vina: повышение скорости и точности стыковки с помощью новой функции подсчета очков, эффективной оптимизации и многопоточности. J Comput Chem. 2009 [бесплатная статья PMC] [PubMed] [Google Scholar]
2009 [бесплатная статья PMC] [PubMed] [Google Scholar]
[105] Bohm HJ. Разработка простой эмпирической оценочной функции для оценки константы связывания комплекса белок-лиганд с известной трехмерной структурой. J Comput Aided Mol Des. 1994;8(3):243–256. [PubMed] [Google Scholar]
[106] Тиг С.Дж. Значение гибкости белков для открытия лекарств. Nat Rev Drug Discov. 2003;2(7):527–541. [PubMed] [Google Scholar]
[107] Gschwend DA, Good AC, Kuntz ID. Молекулярная стыковка на пути к открытию лекарств. Дж Мол Признать. 1996;9(2):175–186. [PubMed] [Google Scholar]
[108] Тотров М., Абагян Р. Стыковка белок-лиганд как проблема оптимизации энергии. В: Раффа РБ, редактор. Термодинамика лекарственного рецептора: введение и экспериментальные применения. Джон Уайли и сыновья; Нью-Йорк: 2001. стр. 603–624. [Академия Google]
[109] Лич АР. Стыковка лиганда с белками с дискретной гибкостью боковой цепи. Дж Мол Биол. 1994; 235(1):345–356. [PubMed] [Google Scholar]
[110] Desmet J, De Maeyer M, Hazes B, Lasters I. Теорема об исключении тупика и ее использование для позиционирования боковой цепи белка. Природа. 1992; 356: 539–542. [PubMed] [Google Scholar]
Теорема об исключении тупика и ее использование для позиционирования боковой цепи белка. Природа. 1992; 356: 539–542. [PubMed] [Google Scholar]
[111] Абагян Р., Тотров М. Смещенная вероятность Конформационные поиски Монте-Карло и электростатические расчеты для пептидов и белков. Дж Мол Биол. 1994;235(3):983–1002. [PubMed] [Google Scholar]
[112] Morris GM, Huey R, Lindstrom W, Sanner MF, Belew RK, Goodsell DS, Olson AJ. AutoDock4 и AutoDockTools4: автоматическая стыковка с избирательной гибкостью рецепторов. J Comput Chem. 2009;30(16):2785–2791. [Бесплатная статья PMC] [PubMed] [Google Scholar]
[113] Knegtel RM, Kuntz ID, Oshiro CM. Молекулярный докинг к ансамблям белковых структур. Дж Мол Биол. 1997;266(2):424–440. [PubMed] [Google Scholar]
[114] Карлсон Х.А., Масукава К.М., Рубинс К., Бушман Ф.Д., Йоргенсен В.Л., Линс Р.Д., Бриггс Дж.М., Маккаммон Дж.А. Разработка динамической модели фармакофора для интегразы ВИЧ-1. J Med Chem. 2000;43(11):2100–2114.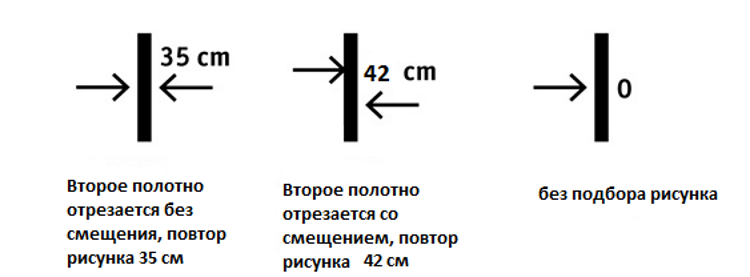 [PubMed] [Академия Google]
[PubMed] [Академия Google]
[115] Кавасотто К.Н., Абагян Р.А. Гибкость белков в стыковке лигандов и виртуальном скрининге на протеинкиназы. Дж Мол Биол. 2004;337(1):209–225. [PubMed] [Google Scholar]
[116] Дерремо П., Шлик Т. Движение открытия/закрытия петли фермента триозофосфатизомеразы. Биофиз Дж. 1998; 74 (1): 72–81. [Бесплатная статья PMC] [PubMed] [Google Scholar]
[117] Zeng L, Zhou MM. Бромодомейн: домен, связывающий ацетиллизин. ФЭБС лат. 2002;513(1):124–128. [PubMed] [Академия Google]
[118] Venkitakrishnan RP, Zaborowski E, McElheny D, Benkovic SJ, Dyson HJ, Wright PE. Конформационные изменения петель активного центра дигидрофолатредуктазы во время каталитического цикла. Биохимия. 2004;43(51):16046–16055. [PubMed] [Google Scholar]
[119] Go N, Scheraga HA. Замыкание кольца и локальные конформационные деформации цепных молекул. Макромолекулы. 1970;3(2):178–187. [Google Scholar]
[120] Додд Л.Р., Бун Т.Д., Теодору Д.Н. Алгоритм согласованного вращения для атомистического моделирования методом Монте-Карло расплавов полимеров и стекол. Мол. физ. 1993;78:961–996. [Google Scholar]
Мол. физ. 1993;78:961–996. [Google Scholar]
[121] Хоффманн Д., Кнапп В. Укладка полипептидов с внерешеточной динамикой Монте-Карло: метод. Евро. Биофиз. Дж. 1996; 111:387–404. [Google Scholar]
[122] Ву М.Г., Дим М.В. Аналитический ребридинг Монте-Карло: применение к цис/транс-изомеризации пролинсодержащих циклических пептидов. Дж. Хим. физ. 1999; 14:6625–6632. [Google Scholar]
[123] Ужин AR. Локальные деформации полимеров с непланарными жесткими внутренними координатами основной цепи. Дж. Комп. хим. 2000; 21:1132–1144. [Академия Google]
[124] Mezei M. Эффективная выборка Монте-Карло для длинных молекулярных цепей с использованием локальных перемещений, протестированная на сольватированном липидном бислое. Дж. Хим. физ. 2003; 118:3874–3879. [Google Scholar]
[125] Cui M, Mezei M, Osman R. Прогнозирование структур белковых петель с использованием подхода Монте-Карло с локальным перемещением и силового поля на основе сетки. Белок Eng Des Sel. 2008;21(12):729–735.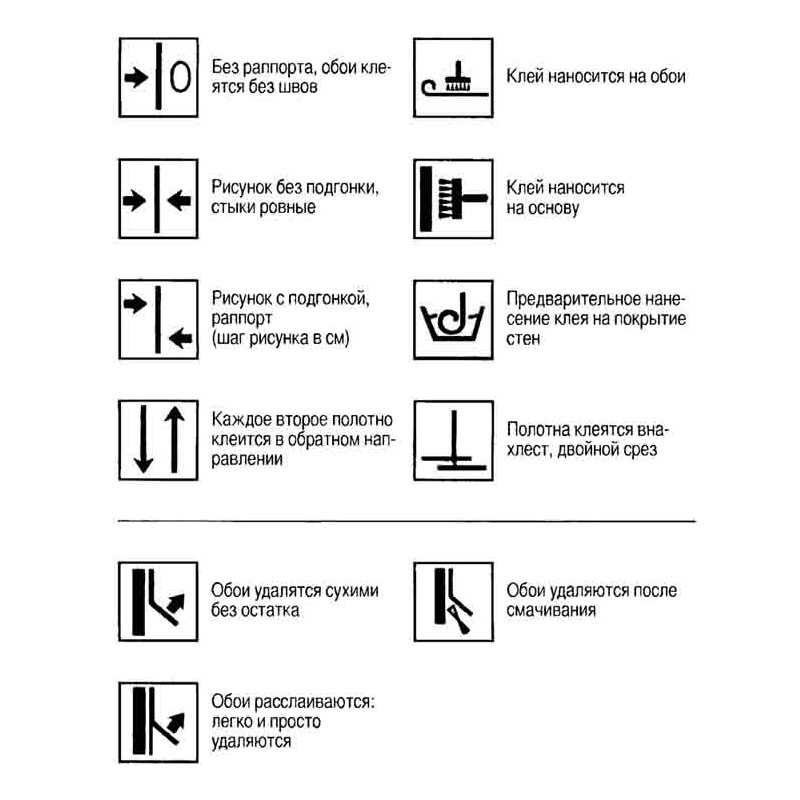 [Бесплатная статья PMC] [PubMed] [Google Scholar]
[Бесплатная статья PMC] [PubMed] [Google Scholar]
[126] Кубиньи Х. Компьютерные приложения в фармацевтических исследованиях и разработках. Джон Уайли; Нью-Йорк: 2006. [Google Scholar] 9.0016
[127] Kroemer RT. Дизайн лекарств на основе структуры: стыковка и оценка. Текущая наука о белках и пептидах. 2007; 8: 312–328. [PubMed] [Google Scholar]
[128] Venhorst J, ter Laak AM, Commandeur JN, Funae Y, Hiroi T, Vermeulen NP. Моделирование гомологии изоформ цитохрома P450 2D (CYP2D) крысы и человека и компьютерная рационализация экспериментальных особенностей связывания лиганда. J Med Chem. 2003;46(1):74–86. [PubMed] [Google Scholar]
[129] Williams PA, Cosme J, Ward A, Angove HC, Matak Vinkovic D, Jhoti H. Кристаллическая структура человеческого цитохрома P450 2C9со связанным варфарином. Природа. 2003; 424 (6947): 464–468. [PubMed] [Google Scholar]
[130] Meng XY, Zheng QC, Zhang HX. Сравнительный анализ сайтов связывания между CYP2C38 и CYP2C39 мыши, основанный на моделировании гомологии, моделировании молекулярной динамики и исследованиях стыковки. Биохим Биофиз Акта. 2009;1794(7):1066–1072. [PubMed] [Google Scholar]
Биохим Биофиз Акта. 2009;1794(7):1066–1072. [PubMed] [Google Scholar]
[131] Boehm HJ, Boehringer M, Bur D, Gmuender H, Huber W, Klaus W, Kostrewa D, Kuehne H, Luebbers T, Meunier-Keller N, Mueller F. Новые ингибиторы ДНК-гираза: предвзятый скрининг иглы на основе трехмерной структуры, проверка попадания биофизическими методами и трехмерная управляемая оптимизация. Многообещающая альтернатива случайному скринингу. J Med Chem. 2000;43(14):2664–2674. [PubMed] [Академия Google]
[132] Киртон С.Б., Мюррей К.В., Вердонк М.Л., Тейлор Р.Д. Прогнозирование способов связывания лигандов в цитохромах Р450 и других гемсодержащих белках. Белки. 2005;58(4):836–844. [PubMed] [Google Scholar]
[133] Доман Т.Н., Макговерн С.Л., Уизерби Б.Дж., Кастен Т.П., Курумбайл Р., Сталлингс В.К., Коннолли Д.Т., Шойхет Б.К. Молекулярный докинг и высокопроизводительный скрининг новых ингибиторов протеинтирозинфосфатазы-1B. J Med Chem. 2002;45(11):2213–2221. [PubMed] [Академия Google]
[134] Шойхет Б. К., Лич А.Р., Кунц И.Д. Сольватация лиганда в молекулярном докинге. Белки. 1999;34(1):4–16. [PubMed] [Google Scholar]
К., Лич А.Р., Кунц И.Д. Сольватация лиганда в молекулярном докинге. Белки. 1999;34(1):4–16. [PubMed] [Google Scholar]
[135] Лорбер Д.М., Шойхет Б.К. Гибкая стыковка лигандов с использованием конформационных ансамблей. Белковая наука. 1998;7(4):938–950. [Бесплатная статья PMC] [PubMed] [Google Scholar]
[136] Freymann DM, Wenck MA, Engel JC, Feng J, Focia PJ, Eakin AE, Craig SP. Эффективная идентификация ингибиторов, нацеленных на конформацию закрытого активного центра HPRT из Trypanosoma cruzi. хим. биол. 2000;7(12):957–968. [PubMed] [Google Scholar]
[137] Su AI, Lorber DM, Weston GS, Baase WA, Matthews BW, Shoichet BK. Стыковка молекул по семействам для увеличения разнообразия совпадений на экранах базы данных: вычислительная стратегия и экспериментальная оценка. Белки. 2001;42(2):279–293. [PubMed] [Google Scholar]
[138] Gschwend DA, Kuntz ID. Новый взгляд на ориентационную выборку и минимизацию твердого тела в молекулярном докинге: оптимизация на лету и устранение вырождения.